Geni associati
Si definiscono geni associati i geni che appartengono ad un gruppo di associazione, ovvero che sono localizzati sullo stesso cromosoma e che non segregano indipendentemente. Infatti poiché è il cromosoma (e non il gene) l'unità che si trasmette durante la meiosi, i geni associati non sono liberi di distribuirsi in modo indipendente gli uni dagli altri e si dice che mostrano associazione o concatenazione[1], o linkage per usare il termine inglese (pron. [ˈlinkiʤ] o [ˈlinkeʤ][2]), negli incroci genetici.
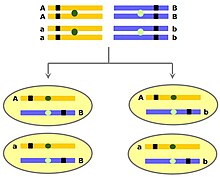
Linkage genetico
modificaSe consideriamo due coppie di cromosomi, ciascuno contenente una coppia di geni in eterozigosi, non si osserva associazione ed i geni si distribuiscono in modo indipendente durante la produzione dei gameti. Infatti quando ha luogo un elevato numero di eventi meiotici, si formano quattro tipi di gameti geneticamente differenti, in uguale proporzione e ciascuno contenente una combinazione diversa degli alleli ai due loci. La situazione cambia se gli stessi geni si trovano sullo stesso cromosoma. Se non avviene alcun crossing-over tra i due geni associati si formano solo due gameti geneticamente diversi: ciascun gamete riceve gli alleli presenti su uno o sull'altro omologo che è stato trasmesso intatto durante la segregazione. Questo caso illustra il cosiddetto linkage completo, che produce solo gameti parentali o non ricombinanti (i due gameti parentali si formano in proporzioni uguali). Se tra i due geni associati avviene crossing-over, ad esempio tra due cromatidi non fratelli dei quattro cromatidi presenti nella tetrade, si vengono a formare due nuove combinazioni alleliche, definite gameti ricombinanti. Questo caso è un esempio di linkage parziale, che produce gameti sia parentali che ricombinanti. Generalmente la frequenza con la quale il crossing-over avviene tra due geni associati è proporzionale alla distanza che separa i rispettivi loci sul cromosoma (distanza interlocus), ciò vuol dire che all'aumentare della distanza tra due geni, la proporzione di gameti ricombinanti aumenta e quella di gameti parentali diminuisce.
Il rapporto di linkage
modificaSe esiste linkage completo tra due geni, a causa della loro stretta vicinanza, e organismi ,eterozigoti ad entrambi i loci, sono incrociati tra di loro, sarà prodotto un unico rapporto fenotipico della F2 che prende il nome di rapporto di linkage. Possiamo spiegare questo rapporto considerando un incrocio che coinvolge i geni mutanti recessivi strettamente associati brown (bw, occhi bruni) e heavy wing vein (hv, venature delle ali ispessite) in Drosophila melanogaster. Gli alleli normali bw+ e hv+, entrambi dominanti, codificano rispettivamente per i caratteri occhi rossi e venature sottili delle ali. In questo incrocio, i moscerini con gli occhi mutati marroni e le venature delle ali normali (sottili) sono incrociati con i moscerini con occhi normali rossi e venature mutante spesse. Poiché i geni sono localizzati su un autosoma non è necessario distinguere tra maschi e femmine.
Nella generazione F1 ciascun moscerino riceve uno dei due cromosomi omologhi da ogni genitore; tutti i moscerini sono eterozigoti per entrambi i geni e mostreranno i tratti dominanti occhi rossi e venature sottili. A causa del linkage completo, quando gli individui della generazione F1 sono incrociati tra loro, ciascun individuo della generazione F1 forma solo gameti parentali. Dopo la fecondazione si produrrà la generazione F2 con un rapporto fenotipico e genotipico 1:2:1.
Un quarto degli individui di questa generazione mostrerà occhi bruni e venature sottili, la metà mostrerà entrambi i caratteri normali ed un quarto mostrerà occhi rossi e venature spesse. Questo rapporto è caratteristico del linkage completo, che è osservabile solo quando due geni sono molto vicini tra loro e la progenie è relativamente piccola. Per fare un confronto possiamo fare un testcross o reincrocio di prova con moscerini della generazione F1. Questo incrocio produce un rapporto 1:1 di moscerini marroni, sottili e rossi, spesse.
Se i geni che controllano questi caratteri fossero in linkage parziale o su autosomi diversi, il testcross avrebbe prodotto quattro, invece di due, fenotipi. Quando si considera un numero elevato di geni mutati in una determinata specie, i geni localizzati sullo stesso cromosoma mostreranno evidenze di associazione tra loro. Di conseguenza, possiamo stabilire un gruppo di linkage, per ciascun cromosoma. In teoria, il numero di gruppo di linkage dovrebbe corrispondere al numero aploide di cromosomi.
Metodo del LOD score per la stima della frequenza di ricombinazione
modificaIl metodo del LOD score (logarithm of odds), sviluppato nel 1955 dal genetista matematico Newton E. Morton,[3] è un test statistico spesso utilizzato per analizzare i rapporti di associazione tra i geni (linkage) in popolazioni animali, umane e vegetali. Il LOD score confronta: (1) la probabilità di riscontrare i valori osservati se i due loci sono effettivamente sullo stesso cromosoma, con una determinata frequenza di ricombinazione, e (2) la probabilità di ottenere gli stessi risultati se i due loci non sono associati, quindi esclusivamente per caso. L'analisi computerizzata del LOD score è un metodo semplice per analizzare alberi genealogici complessi e determinare il linkage tra i tratti mendeliani (o tra un tratto e un marker, o due marker). Una descrizione[4] sommaria del suo funzionamento è:
- Costruzione di un albero genealogico;
- Stimare la frequenza di ricombinazione;
- Calcolare il LOD score per ognuna delle stime;
- La stima con il LOD score massimo sarà considerata la migliore.
Il LOD score è calcolato come:
NR denota il numero di prole non-ricombinante, ed R denota il numero di prole ricombinante. Il motivo per cui si utilizza 0.5 al denominatore è che ogni allele che sia completamente unlinked (e.g. alleli su cromosomi distinti) ha il 50% di possibilità di ricombinare a causa dell'assortimento indipendente.
Teta è la frazione ricombinante, ed è uguale a R / (NR + R) . I risultati vengono espressi come log 10 del rapporto delle due probabilità e per convenzione, un LOD score più grande di 3.0 è considerato prova di linkage, infatti un LOD score di +3 indica una probabilità 1000:1 che il linkage osservato non avvenga per caso. Mentre un LOD score più basso di -2.0 è considerato sufficiente per escludere la probabilità di linkage. Sebbene sia assai poco probabile che un LOD score di 3 sia ottenuto a partire da un singolo albero genealogico, le proprietà matematiche del test permettono di combinare le informazioni derivanti da più alberi sommando i singoli LOD score. È importante ricordare che la soglia tradizionale di LOD > +3 è arbitraria e che in alcuni casi, in particolare analisi di tratti genetici complessi con centinaia di marker, il valore massimo dovrebbe essere aumentato.
Note
modifica- ^ Concatenazione, in Treccani.it – Vocabolario Treccani on line, Roma, Istituto dell'Enciclopedia Italiana. URL consultato il 13 ottobre 2016.«In genetica, termine usato talora come sinon. di associazione genica, per traduzione dell’ingl. linkage.»
- ^ Luciano Canepari, linkage, in Il DiPI: dizionario di pronuncia italiana, Bologna, Zanichelli, 1999, ISBN 88-08-09344-1.
- ^ Morton NE, Sequential tests for the detection of linkage, in American Journal of Human Genetics, vol. 7, n. 3, 1955, pp. 277–318, PMC 1716611, PMID 13258560.
- ^ Il metodo è descritto in gran dettaglio da Strachan e Read
Bibliografia
modifica- Klug William S., Kummings Micheal R., Spencer Charlotte A., Concetti di genetica, Pearson, 2007 ISBN 8871923189
- Russell Peter J., i-genetica, Edises, 2007 ISBN 8879593854
- Russell Peter J., Genetica. Un approccio molecolare (3ª edizione), Pearson, 2010
Voci correlate
modificaAltri progetti
modifica- Wikimedia Commons contiene immagini o altri file su Geni associati
| Controllo di autorità | LCCN (EN) sh85077241 · GND (DE) 4197281-8 · J9U (EN, HE) 987007529214405171 |
|---|